por CenterLab | jan 9, 2020 | Informativos
Estudo identifica uma assinatura de 16 espécies microbianas capaz de predizer o câncer colorretal; resultados podem contribuir para criar exame preditivo válido para populações com diferentes culturas alimentares.
O microbioma, conjunto de microrganismos presentes no intestino, pode ser usado para prever a ocorrência do câncer colorretal – o segundo tipo de tumor mais frequente em mulheres e o terceiro entre os homens.
Uma pesquisa feita por uma equipe internacional, com participação brasileira, identificou padrões no microbioma intestinal – que independem da cultura alimentar das populações estudadas – e detectou associação entre alterações nesse padrão e a ocorrência de câncer colorretal. A descoberta abre caminho para o desenvolvimento de exames não invasivos capazes de prever a ocorrência da doença.
O estudo, publicado nesta segunda-feira (01/04) na revista Nature Medicine, tem como primeiro autor Andrew Maltez Thomas, doutor em Bioinformática pela Universidade de São Paulo (USP), e teve apoio da FAPESP por meio de uma Bolsa de Pesquisa no Exterior, realizada na Universidade de Trento, na Itália.
Os pesquisadores combinaram análise de metagenômica, bioinformática e aprendizagem de máquina (com uso de inteligência artificial) para correlacionar a ocorrência do câncer colorretal com dados do microbioma de 969 pessoas da Alemanha, França, Itália, China, Japão, Canadá e Estados Unidos. Trata-se de um dos maiores e mais variados estudos sobre o tema.
Os resultados não só estabelecem conjuntos de microrganismos associados ao câncer colorretal em todas as populações estudadas, mas também indicam assinaturas no metabolismo microbiano (padrão de metabólitos produzidos pelos microrganismos) que também têm poder de prever a ocorrência da doença.
A análise rendeu ainda outros dois achados importantes. Um deles se refere a maior prevalência de bactérias comumente encontradas na boca e nas vias aéreas no intestino de pacientes com câncer colorretal. A outra descoberta indica a associação entre a doença e a presença de uma enzima microbiana que degrada a colina, um nutriente que faz parte do complexo B de vitaminas.
No estudo, pacientes com câncer colorretal apresentaram maior presença de bactérias da espécie Fusobacterium nucleatum em comparação com indivíduos saudáveis. Essa bactéria normalmente habita regiões da boca. Era esperado, até então, que o ambiente ácido do estômago fosse fatal para tais microrganismos.
“Há um aporte maior de espécies orais indo para o intestino em pacientes com câncer colorretal. Talvez essa migração cause inflamações no intestino, originando o tumor. No entanto, não sabemos ainda o real motivo de elas se transportarem para o intestino. Sabemos apenas que há uma associação entre a sua presença no intestino e o câncer colorretal. É algo que ainda precisa ser melhor entendido”, disse Thomas.
A detecção de maior abundância do gene da enzima microbiana colina-trimetil-liase (cutC) nas amostras fecais dos pacientes com câncer mostra uma possível ligação entre a microbiota e os resultados de estudos anteriores sobre a relação da doença com uma alimentação rica em gordura.
“Essa enzima degrada a colina, metabólito presente em dietas com altas concentrações de carne vermelha e de outros alimentos gordurosos. Após degradar a colina ela libera acetaldeído, uma conhecida substância carcinogênica”, disse Thomas.
No estudo, os pesquisadores analisaram dados sobre a composição e a abundância de todas as bactérias encontradas nas 969 amostras fecais. Para obter um método de análise mais simples, que possa ser usado amplamente em clínicas e hospitais, os pesquisadores conseguiram selecionar as bactérias com maior peso na análise.
“Com 16 espécies, obtivemos resultados comparáveis às análises feitas com todas as espécies. É um passo, portanto, na direção de se ter uma ferramenta diagnóstica simples, sem precisar sequenciar toda a microbiota e sem deixar de ter a precisão necessária”, disse Thomas.
Causa ou consequência
A relação entre microbiota do intestino e saúde humana é uma área de pesquisa que tem crescido, sobretudo nos últimos 10 anos. O novo estudo, no entanto, segue um conceito inovador de utilização de bactérias como marcadores de desenvolvimento de uma doença.
“O mais comum é buscar marcadores associados diretamente a células tumorais. Em nosso trabalho, o conceito é outro. A análise é feita a partir de alterações de um conjunto relativamente pequeno de bactérias dentro de um espectro de centenas de bactérias que estão no intestino e que podem indicar uma doença”, disse Emmanuel Dias-Neto, do Centro Internacional de Pesquisas (Cipe) no A.C.Camargo Cancer Center, também autor do artigo.
Com a análise de sequências de DNA obtidas da microbiota é possível descobrir quais bactérias estão presentes na microbiota de cada amostra, além de identificar a quantidade de cada uma das bactérias e variantes do genoma desses microrganismos que podem estar relacionadas com diferentes desfechos – como, por exemplo, a ocorrência ou risco aumentado de câncer colorretal.
No entanto, é preciso destacar que o estudo não revelou que uma microbiota alterada causa o câncer colorretal.
“Foi detectada uma associação, o que não implica necessariamente uma relação causal. A pergunta que fica é: são essas determinadas bactérias que estão provocando o câncer ou é o câncer que cria um ambiente diferente no duto colorretal e, assim, faz com que certas bactérias sejam favorecidas em relação a outras? Ainda não temos essa resposta, que será fundamental para que os resultados deste artigo possam futuramente ajudar no desenvolvimento de terapias para o tratamento do câncer colorretal”, disse João Carlos Setubal, coordenador do Programa Interunidades de Pós-Graduação em Bioinformática da USP e outro autor do artigo. Setubal e Dias-Neto orientaram o doutorado de Thomas.
Análise computacional
Segundo os pesquisadores, esta é talvez a maior análise sobre câncer colorretal com dados de amostras fecais e com populações tão diversas. A equipe de pesquisadores analisou os dados de cinco estudos públicos com dados de outros dois estudos realizados pelos pesquisadores na Universidade de Trento.
Com as informações obtidas nos sete estudos, foi possível identificar enzimas, bactérias e como a microbiota é capaz de predizer a presença do câncer colorretal. Os dados de outros dois estudos, com mais 200 amostras, foram usados para validar os achados.
“O sequenciamento de DNA das amostras – cuja análise exige distinguir entre DNA da microbiota e DNA humano – foi uma forma de identificar e quantificar as espécies de microrganismos e seus genes presentes nessas amostras. Extraímos o DNA das amostras fecais e o sequenciamos. Depois, com métodos computacionais, analisamos os dados. Conseguimos identificar e quantificar quais espécies estavam presentes e qual era a abundância dos genes”, disse Thomas.
Por serem dados oriundos de estudos diferentes, os pesquisadores usaram métodos estatísticos sofisticados para analisá-los em conjunto.
“Empregamos análises estatísticas que são usadas para fazer meta-análise, e também foram utilizadas técnicas de aprendizado de máquina para compreendermos o quão preditivos são os resultados”, disse Thomas.
Além de validar os dados, o grupo de Nicola Segata, da Universidade de Trento e líder do projeto, teve seus resultados reforçados por outro estudo realizado no European Molecular Biology Laboratory (EMBL), com sede na Alemanha. Os cientistas do EMBL também estudaram a relação entre microbioma e câncer e tiveram artigo publicado na mesma edição da Nature Medicine.
“Ao longo da preparação dos artigos, trocamos dados e informações com o outro grupo, numa parceria que se mostrou muito importante para reforçar nossos achados. Apesar de usarmos técnicas de aprendizagem de máquina e métodos estatísticos diferentes chegamos ao mesmo resultado: que o microbioma intestinal é capaz de predizer a presença de câncer colorretal em diferentes populações e estudos”, disse Thomas. Com informações da Fapesp.
Fonte: Labnetwork
Publicado em: 02/04/2019

por CenterLab | jan 7, 2020 | Informativos
A resistência aos antimicrobianos é considerada um dos desafios aos sistemas de saúde atuais. Estima-se que 700 mil mortes sejam causadas anualmente pela resistência aos antimicrobianos. De acordo com essas análises e sem a mudança de abordagem no âmbito geral para conter esse problema, estimasse que até 2050 esse número possa superar as mortes por decorrência do câncer.
A Organização Mundial de Saúde (OMS) define Resistência aos antimicrobianos como a “capacidade de um microorganismo impedir a atuação de um antimicrobiano”. Tornando o tratamento ineficaz, e as infecções persistentes e até incuráveis. Em 2019 a OMS apontou o tema como a quinta ameaça à saúde global.
Segundo a OMS, para evitar que isso ocorra, precisamos investir em múltiplas frentes, como por exemplo: medidas de prevenções pela lavagem correta das mãos e aplicações de vacinas (quando houver), pesquisa e produção de medicamentos mais eficazes, testes rápidos para identificar o agente infeccioso como forma de impedir prescrições desnecessárias e ainda alertar a população sobre os perigos da automedicação e de interromper o tratamento ao primeiro sinal de melhora.
No Brasil, de acordo com a Agência Nacional de Vigilância Sanitária (ANVISA), aproximadamente 25% das infecções registradas são causadas por microorganimos multirresistentes, ou seja, aqueles que se tornam imunes à ação dos antimicrobianos.
AS BACTÉRIAS:
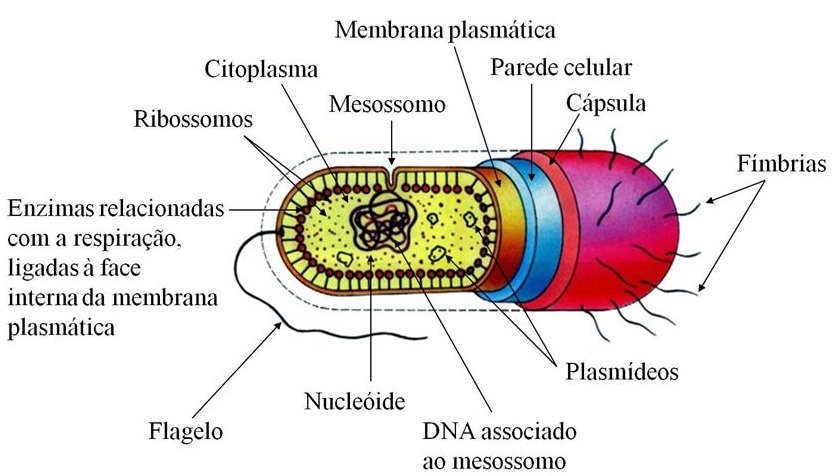
As bactérias de forma bem simples podem ser descritas como: organismos unicelulares e procarióticos (não possui membrana nuclear), ou seja, o material genético fica disperso no citoplasma sendo composto por DNA de cadeia única e circular em dupla hélice e podem apresentar estruturas como membrana plasmática, cápsulas, esporos, parede celular, flagelos além dos plasmídeos, segue ao lado, figura ilustrativa.
SUPERBACTÉRIAS:
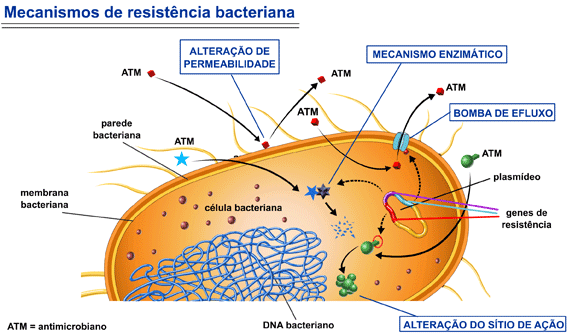
As “Superbactérias” ou bactérias multirresistentes são as bactérias que possuem resistência a vários antibióticos, fato esse que dificulta o tratamento das infecções. Esse mecanismo de resistência bacteriana pode ocorrer por diversas formas os principais são:
– Alteração na permeabilidade da membrana bacteriana;
– Alteração no sitio de atuação do antibiótico;
– Bomba de efluxo: bombeamento ativo do antibiótico para o exterior do citoplasma;
– Produção de enzimas que eliminam os antibióticos, esses mecanismo destaca-se como sendo a estratégia mais frequente observadas nas superbactérias, por esse modo trataremos com destaque esse mecanismo a seguir.
Com frequência bactérias utilizam mais de uma estratégia para evitar a ação dos antimicrobianos; assim, a ação conjunta de múltiplos mecanismos pode produzir um aumento acentuado da resistência aos antimicrobianos.
ANTIBIOTICOS E CLASSIFICAÇÃO:
Os antibióticos podem ser definidos de uma forma bem geral como compostos naturais – quando produzidos por fungos ou bactérias – ou sintéticos que são capazes de interferir no crescimento bacteriano (bacterostáticos) ou causar a morte dos mesmos (bactericidas).
Os antibióticos de origem natural e seus derivados semi-sintéticos compreendem a maioria dos antibióticos de uso clínico e podem ser classificados em β-lactâmicos (penicilinas, cefalosporinas, cabapeninas, oxapeninas e monobactamas), as tetraciclinas, os aminoglicosídeos, macrolídeos, peptídicos cíclicos (glicopeptídeos, lipodepsipeptídeos), estreptograminas, entre outros (lincosaminas, clorafenicol, rifamicinas etc). Os de origem sintética são classificados em sulfonamidas, fluoroquinolonas e oxazolidinonas
MECANISMO RESISTÊNCIA ENZIMÁTICA:
O mecanismo de resistência bacteriano mais importante e frequente é a degradação do antimicrobiano por enzimas. As β- lactamases hidrolisam a ligação amida do anel beta-lactâmico, destruindo, assim o sítio de ligação. São descritas numerosas β- lactamases diferentes. Essas enzimas são codificadas em cromossomos ou sítios extracromossômicos através de plasmídeos ou transposons, podendo ser produzidas de modo constitutivo ou ser induzido.
Nas bactérias Gram-negativas, o papel das β-lactamases na resistência é complexo e extenso. Verifica-se a presença de quantidades abundantes de enzimas; muitas delas inativam vários antimicrobianos β-lactâmicos, e os genes que codificam essas β- lactamases estão sujeitos a mutações que expandem a atividade enzimática e que são transferidos de modo relativamente fácil. Além disso, as β-lactamases de bactérias Gram-negativas são secretadas no espaço periplasmático, aonde atuam em conjunto com a barreira de permeabilidade da parede celular externa, produzindo resistências clinicamente significativas a antimicrobianos.
– β-lactamases de espectro estendio (ESBLs):
São enzimas que hidrolisam a maioria dos β-lactâmicos poupando apenas as cefaminas (como cefoxitina) e carbapenêmicos. A epidemiologia de ESBLs entre Enterobacteriaceae mudou recentemente com a disseminação difundida de enzimas do tipo CTX-Ms nas espécies de Escherichia coli. Em qualquer lugar do mundo, a CTX-Ms representa a maioria das ESBLs, a ponto de sua difusão ser qualificada como pandêmica. Eles estão igualmente isolados em ambientes comunitários e hospitalares e parecem ser endêmicos em instituição de cuidados de longo prazo. A crescente prevalência de ESBLs no ambiente da comunidade cria um problema sem precedentes.
– Carbapenemases:
São enzimas capazes de hidrolisar todos os β-lactâmicos (penicilinas, cefalosporinas, aztreonam, carbapenêmicos), classe de antimicrobianos mais diversificada e largamente utilizada. Ressaltando ainda, que nestes casos, aminoglicosídeos e fluoroquinolonas apresentam, muitas vezes, sensibilidade diminuída, deixando a antibioticoterapia bastante reduzida.
Dentre essas carbapenemases, a β-lactamases KPC (classe A) espalhou-se pelo mundo na década de 2000. Depois disto, também se tem observado a expansão das metalo-β-lactamases (MBL) do tipo IMP, VIM e NDM (Classe B), bem como das OXA-48 (Classe D). Estas são principalmente detectadas em ambientes hospitalares e são responsáveis pela maior parte das infeções nasocomiais e causam um grande problema em termos de saúde global, especialmente pelo fato da sua presença ser difícil de detectar. Lembrando que a identificação dessa enzima interfere diretamente no tratamento do paciente.
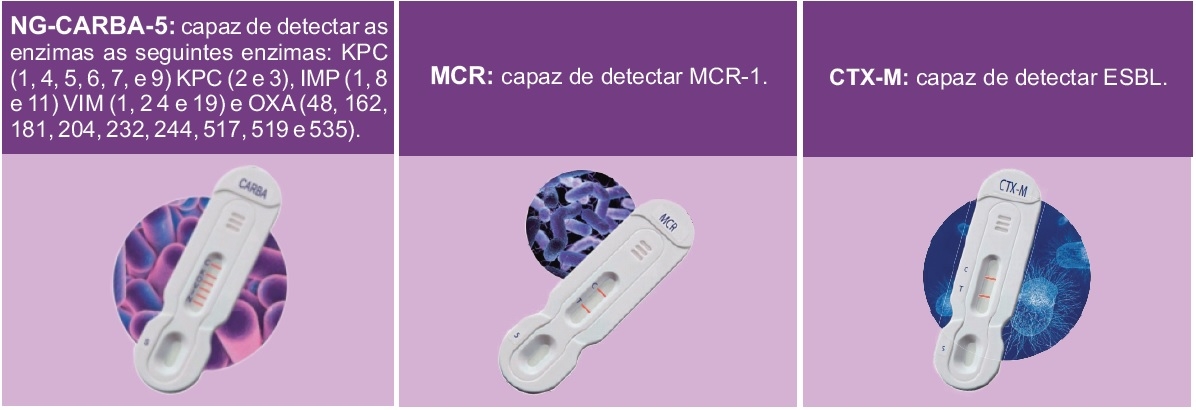
Em decorrência desse grande problema de saúde publica e preocupação com os pacientes a Laborclin em parceria com a Centerlab tem a satisfação de apresentar a todos nossos clientes, parceiros e profissionais de saúde uma grande inovação no diagnóstico dessas enzimas através dos testes NG-CARBA 5, MCR e CTX-M.
Tratam-se de testes Imunogromatográficos com detecção dessas enzimas a partir da colônia bactéria ou hemoculturas com resultados em 15 minutos e com excelente correlação ao PCR (Reação de Polimerase em Cadeia), com pouca necessidade de treinamento, não sendo necessária a utilização de equipamentos e eliminando custos de manutenção. Além disso os kits estáveis à temperatura ambiente.
Nosso intuito com esse informativo é alertar aos profissionais de saúde e hospitais apresentando inovações e tecnologias disponíveis para um diagnóstico preciso, rápido e eficaz auxiliando assim o tratamento dos pacientes. Para se aprofundar nos estudos recomendamos uma leitura mais ampla e completa dos tópicos em questão.
Referências:
– https://www.laborclin.com.br/wp-content/uploads/2019/10/646001-NG-TEST-CARBA-5-KIT-20T.pdf
– https://www.laborclin.com.br/wp-content/uploads/2019/12/646005-%E2%80%93-NG-TEST-CTX-M-%E2%80%93-KIT-20T.pdf
– https://www.laborclin.com.br/wp-content/uploads/2019/12/646002-NG-TEST-CTX-M-MULTI-KIT-20T.pdf
– https://www.laborclin.com.br/wp-content/uploads/2019/09/NG_CARBA.pdf
– https://www.youtube.com/watch?v=oicMSk4dmsk
– https://portalarquivos2.saude.gov.br/images/pdf/2018/outubro/22/18_Tatiana_Estrela.pdf
– https://www.paho.org/bra/index.php?option=com_content&view=article&id=5664:folha-informativa-resistencia-aos-antibioticos&Itemid=812
– http://www.scielo.br/scielo.php?script=sci_arttext&pid=S0100-40422010000300035
– https://pt.wikipedia.org/wiki/Bact%C3%A9rias
– http://faef.revista.inf.br/imagens_arquivos/arquivos_destaque/UuBDHbHjev9rGKV_2013-6-21-11-52-49.pdf
– http://www.anvisa.gov.br/servicosaude/controle/rede_rm/cursos/rm_controle/opas_web/modulo3/mec_enzimatico.htm
– http://www.ciencianews.com.br/arquivos/ACET/IMAGENS/Noticias_ACET/noticia_1_carbapenemases.pdf




