O uso de métodos moleculares não dependem do isolamento ou crescimento do patógeno ou da detecção de uma resposta imune contra o agente. E sim, da informação genética contida nas amostras. Por isso, o diagnóstico molecular está avançando muito na investigação de doenças infecciosas, genéticas e oncológicas.
O que permitiu esse crescimento foi, principalmente, a expansão do conhecimento sobre os genomas dos vírus, bactérias e fungos aliados aos avanços na bioinformática, que permitiram análises das sequências de ácidos nucléicos para encontrar as melhores regiões para diagnóstico.
Dentre as técnicas descritas na literatura, uma de maior destaque é a reação em cadeia da polimerase (polymerase chain reaction, PCR). Essa técnica é utilizada para a amplificação seletiva de determinada região de uma molécula-alvo de DNA, na qual apenas uma única molécula de DNA pode servir de molde para amplificação produzir milhares de cópias.
Apesar da sensibilidade da PCR ser o diferencial da técnica, alguns fatores podem comprometer o resultado. A contaminação é uma preocupação frequente. As moléculas de DNA previamente amplificadas em outras reações (amplicons) são as que representam as maiores ameaças de contaminação. Por esse motivo, é imprescindível adotar práticas que assegurem sua eficiência e confiabilidade dos resultados, principalmente quando empregada para fins de diagnóstico. A qualidade e a quantidade dos ácidos nucleicos extraídos são bastante afetadas pela coleta da amostra, por seu manuseio e transporte e pela escolha do método de extração. Assim, torna-se relevante entender as possíveis causas de erro, na fase pré-analítica, mais frequentes no diagnóstico molecular.
Amostras de sangue e aspirado de medula óssea
Estudos apontam que a heparina e o heme são potentes inibidores da reação em cadeia da polimerase (PCR), dessa forma os anticoagulantes recomendados para essas amostras são os ácidos etilenodiaminotetracético (EDTA) e citrato dextrose (ACD).
O sangue total é estável a temperatura ambiente por 24 horas para análise de DNA e até oito dias, quando resfriado (2-8°C). Para análise de RNA celular, o sangue deve ser coletado com aditivo estabilizador. A saber, coleta e armazenamento de sangue total sem estabilizador não são recomendados para análise de transcrição genética, em função da indução e da degradação de RNA que ocorre ex vivo.
A medula óssea deve ser coletada utilizando-se uma seringa com EDTA, e a equipe responsável pelo processamento deve ser avisada assim que a amostra chegar ao laboratório. Para extração de DNA, o aspirado de medula óssea pode ser armazenado temporariamente por até 72 horas a 2-8°C antes do processamento. No entanto, caso seja necessário armazenar por tempo superior a esse, devem-se remover os eritrócitos e congelar a amostra a -20°C (com validade de vários meses).
Atenção: a remoção dos eritrócitos pode liberar heme e inibir a reação de PCR
AMOSTRA DE LÍQUIDO CEFALORRAQUIDIANO (LCR)
Amostras de LCR devem ser transportadas a 2-8°C para análise de DNA. Caso não possam ser processadas imediatamente, elas podem ser congeladas (a -20°C ou inferior) para pesquisa de vírus de DNA (HSV, CMV e VZV).
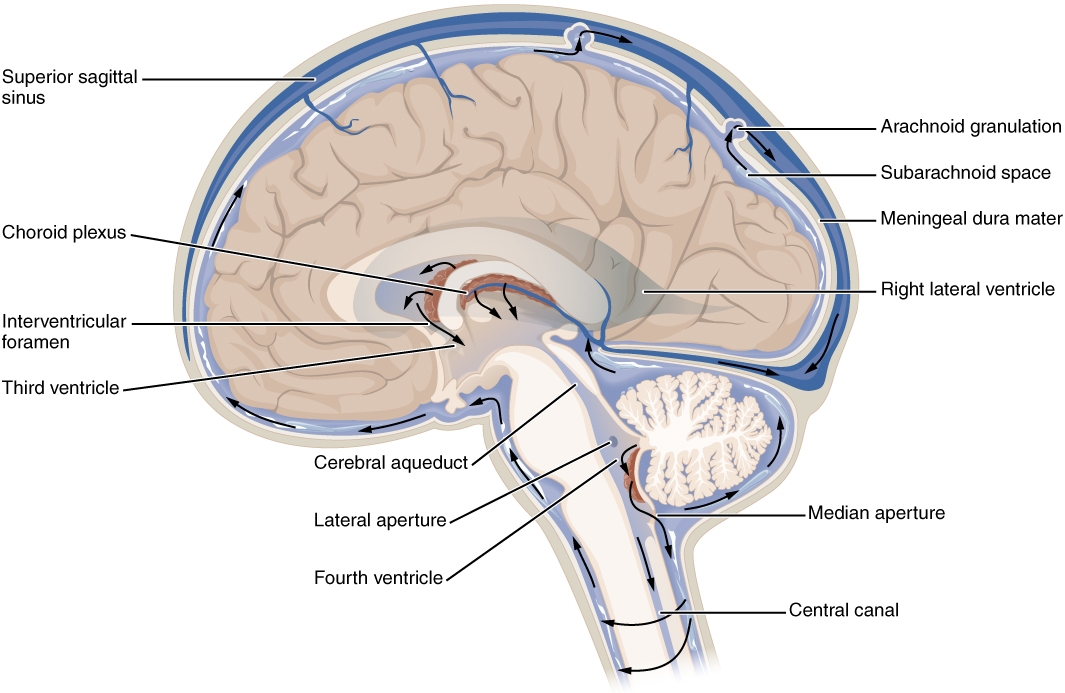
Imagem retirada do site da Kasvi. Notícia publicada em: 20.dez.2019
Para análise de RNA (incluindo vírus de RNA, como os enterovírus), a amostra deve ser colocada em banho com gelo triturado e o RNA extraído em até quatro horas da coleta. Se não for possível, deve-se remover possível contaminação com eritrócitos e congelar a amostra, transportando-a com gelo seco.
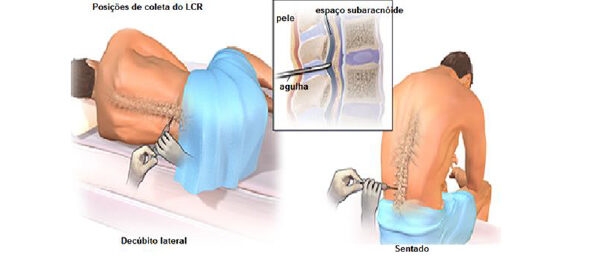
Imagem retirada do site da Kasvi. Notícia publicada em: 20.dez.2019
AMOSTRAS DE ESCARRO
O escarro para análise de DNA deve ser coletado em frasco estéril e transportado ao laboratório à temperatura ambiente, caso demore até 30 minutos; caso contrário, deve ser refrigerado. O DNA no escarro pode ser estável por até um ano quando congelado a -70° C. Alguns protocolos de extração de DNA utilizam a concentração do escarro para aumentar a sensibilidade da pesquisa de agentes infecciosos, e as recomendações do fabricante devem ser seguidas nesses casos.